研究者紹介
ライフサイエンス/計算・解析グループ
[教授] 石川 拓司 [教授] 梅津 光央 [教授] 菊川 豪太 [准教授] 吉留 崇

東北大学 大学院医工学研究科
教授
石川 拓司
バイオメカニクス、計算力学、アクティブマター
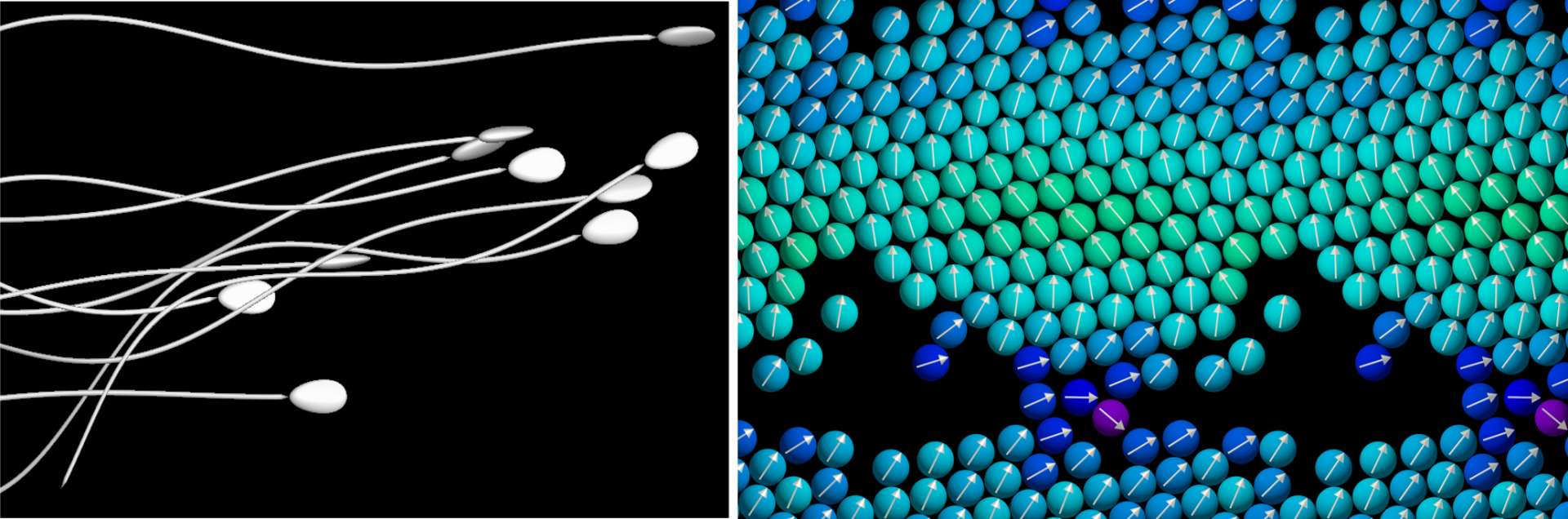
我々は、力学を基盤として生物の機能を解き明かす「バイオメカニクス」の分野を開拓しています。近年、健康問題や環境問題に力学が重要な役割を果たすことが分かってきました。我々はこうした諸問題の解決を目指し、実験と理論、数値シミュレーションを融合したバイオメカニクスを用いて生体機能を解明しています。研究対象は、循環器系・呼吸器系・消化器系の流れや、精子の遊泳と受精、腸内フローラの形成、医療機器開発などの健康問題のみならず、バイオフィルムの形成や赤潮の発生、多細胞動物の進化など、多岐にわたります。
代表的な論文
Cilia and centrosomes: Ultrastructural and mechanical perspectives
Takuji Ishikawa, Hironori Ueno, Toshihiro Omori, Kenji Kikuchi; Seminars in Cell and Developmental Biology 110 (2021) 61–69
真核細胞の繊毛と中心体を対象とし、分子スケールの微細構造と運動、組織スケールでの生物学的機能を、力学的視点から解説した総説である。左右非対称の器官形成過程で重要なノード繊毛や、クリアランス機能で重要な気道繊毛を例として紹介している。
もっと見る
Bacterial biomechanics—From individual behaviors to biofilm and the gut flora
Takuji Ishikawa, Toshihiro Omori, Kenji Kikuchi; APL Bioeng. 4, 041504 (2020); doi: 10.1063/5.0026953
バクテリアを対象とし、さまざまな環境での遊泳運動や懸濁液特性などを力学的視点から解説した総説である。流れ中のバイオフィルムの成長過程や、ゼブラフィッシュ腸内の細菌挙動の数値シミュレーション例を紹介している。
Swimming microorganisms acquire optimal efficiency with multiple cilia
Toshihiro Omoria, Hiroaki Itoa, Takuji Ishikawa; PNAS | December 1, 2020 | vol. 117 | no. 48 | 30201–30207
繊毛で遊泳する微生物の数理モデル化を行い、遊泳のエネルギー効率を数値シミュレーションで求めた。その結果、体長の2乗に比例して繊毛数が増えると最も効率的であることが明らかとなった。この傾向は、自然界の微生物で広く認められた。

東北大学 大学院工学研究科
教授
梅津 光央
機械学習を組み入れた進化分子工学による機能タンパク質の開発
自然界の進化工程を試験管内で模倣した進化分子工学に機械学習などのAI技術を取り入れることによって、目的機能を持つタンパク質を確実・迅速に創出するローリスク・ハイリターン技術の開発を行っている。このプラットフォーム基盤技術を用いて、抗体や酵素を中心として医療・環境・材料分野で活躍できる機能タンパク質を設計し、さらにナノテクノロジーも利用することでアミノ酸だけでは創出できない機能素子を開発している。
代表的な論文
Machine-learning-guided library design cycle for directed evolution of enzymes: the effects of training data composition on sequence space exploration
Yutaka Saito, Misaki Oikawa, Takumi Sato, Hikaru Nakazawa, Tomoyuki Ito, Tomoshi Kameda, Koji Tsuda, and Mitsuo Umetsu; ACS Catalysis, 11(23), 14615–14624 (2021). DOI: org/10.1021/acscatal.1c03753.
酵素タンパク質のアミノ酸配列に変異を加えて、そのアミノ酸配列と酵素の触媒活性の紐づけデータを作成して機械学習にかけることにより、酵素の機能を4倍程度向上した論文です。
もっと見る
Enzymatic ligation of an antibody and arginine 9 peptide for efficient and cell-specific siRNA delivery
Yu Ando, Hikaru Nakazawa, Daisuke Miura, Kaho Otake, and Mitsuo Umetsu; Scientific Reports, 11, 21882 (2021). 2021.11, DOI: org/10.1038/s41598-021-01331-1.
抗体分子に核酸と相互作用するペプチドを融合させた分子を作製することにより、特定細胞にsiRNAを導入できるようにした論文です。
Machine-Learning-Guided Mutagenesis for Directed Evolution of Fluorescent Proteins
Yutaka Saito, Misaki Oikawa, Hikaru Nakazawa, Teppei Niide, Tomoshi Kameda, Koji Tsuda, and Mitsuo Umetsu; ACS Synthetic Biology, 7(9), 2014–2022 (2018). DOI: 10.1021/acssynbio.8b00155
緑色蛍光タンパク質のアミノ酸配列に変異を加えて、そのアミノ酸配列と蛍光特性の紐づけデータを作成して機械学習にかけることにより、黄色に強く光るタンパク質を作製した論文です。

東北大学 流体科学研究所
教授
菊川 豪太
有機・高分子材料における熱物質輸送特性・材料物性のマルチスケール解析
ナノスケールからマクロスケールに渡る多くの工業・産業プロセスにおいては、分子レベルの物理が複合的に関与する現象が数多く見られます。そこで、有機分子膜、高分子材料、ナノスケール構造内の流動などを対象とし、分子動力学法を起点とした大規模数値シミュレーション、マルチスケール数値解析技法および機械学習技術・インフォマティクスの統合によって、ミクロスケールの熱・物質輸送現象およびマクロな材料物性を支配するミクロスケールメカニズムの解明を目指しています。
代表的な論文
A molecular dynamics study on heat transfer characteristics at the interfaces of alkanethiolate self-assembled monolayer and organic solvent
Gota Kikugawa, Taku Ohara, Toru Kawaguchi, Eiichi Torigoe, Yasumasa Hagiwara, Yoichiro Matsumoto; Journal of Chemical Physics, Vol.130 (2009), 074706.
有機分子を1分子層固体表面に修飾した自己組織化単分子膜(SAM)によって、固体と液体の間の熱輸送が促進されることを分子動力学シミュレーションを用いて世界に先駆けて明らかにした。
もっと見る
Effect of crosslink formation on heat conduction in amorphous polymers
Gota Kikugawa, Tapan G. Desai, Pawel Keblinski, and Taku Ohara; Journal of Applied Physics, Vol. 114 (2013), 034302.
高分子材料内部に架橋を導入した際の熱伝導率の変化を調べ、熱伝導率変化の程度が材料に依存することを示した。
Data analysis of multi-dimensional thermophysical properties of liquid substances based on clustering approach of machine learning
Gota Kikugawa, Yuta Nishimura, Koji Shimoyama, Taku Ohara, Tomonaga Okabe, and Fumio S. Ohuchi; Chemical Physics Letters, Vol. 728 (2019), pp. 109-114.
自己組織化マップ(SOM)機械学習を有機液体材料のグルーピングに適用し、データマイニングが材料開発に有効であることを示した。

東北大学 大学院工学研究科 応用物理学専攻
准教授
吉留 崇
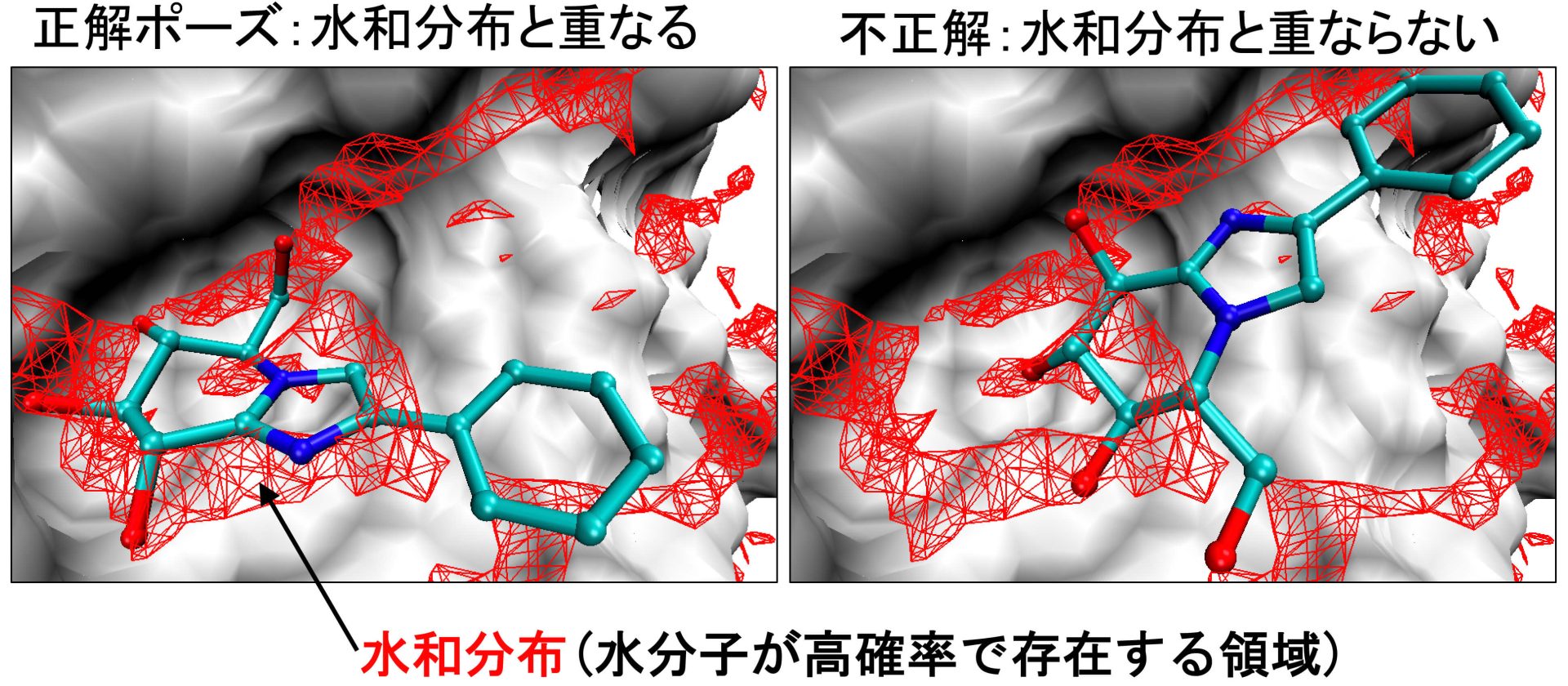
水和に主眼を置いたタンパク質の研究
創薬への応用を目指し、既存のリガンド結合予測(タンパク質に特異的に結合し、その機能を制御する小分子の結合位置を予測すること)では無視されていた「水和」を顕に考慮することで、リガンド結合予測精度を向上させることを目指している。そのためには、タンパク質周りの水の分布(水和分布)を短時間に計算する必要があるが、最近、深層学習を用いて、水和分布を既存の手法の約1000分の1の計算時間(数十秒程度)で得ることに成功した。この手法を武器として、高精度なリガンド結合予測法の確立を目指している。
代表的な論文
Comprehensive 3D-RISM Analysis of the Hydration of Small Molecule Binding Sites in Ligand-Free Protein Structures
T. Yoshidome, M. Ikeguchi, and M. Ohta; J. Comput. Chem., 41, 2406 (2020).
タンパク質の周りの水の性質を包括的に解析した。その結果、水の性質を利用すると、薬剤(リガンド)がタンパク質にどのように結合するかが明らかになる可能性があることが分かった。
もっと見る
Classification of Projection Images of Proteins with Structural Polymorphism by Manifold: A Simulation Study for X-Ray Free-Electron Laser Diffraction Imaging
T. Yoshidome, T. Oroguchi, M. Nakasako, and M. Ikeguchi; Phys. Rev. E, 92, 032710 (2015).
結晶化を行わずに構造解析を行う手法で得られたタンパク質の実験データ(2次元投影像)を、タンパク質の構造変化と投影方向の両面から正しく並べ替えることが出来るプログラム「閻魔」を開発した。




